G7-1 スーパーコンピュータTSUBAME3の計算機利用とインシリコスクリーニング支援
スーパーコンピュータ利用支援、天然変性領域予測支援、大規模PPI予測支援、分子動力学シミュレーションの実施・解析支援、機械学習に関する支援
[1] 支援担当者
| 所属 |
①東京工業大学 科学技術創成研究院 ②東京工業大学 情報理工学院 |
|
|---|---|---|
| 氏名 |
①関嶋 政和 ②秋山 泰、石田 貴士、大上 雅史 |
|
| AMED 事業 |
ユニット/領域名 課題名 |
インシリコユニット 大規模分子シミュレーションによるインシリコスクリーニング支援と構造インフォマティクス技術の高度化 |
| 代表機関 代表者 |
東京工業大学 関嶋 政和 |
|
| 支援技術のキーワード | スーパーコンピュータ、インシリコスクリーニング、蛋白質間相互作用解析、天然変性領域予測、機械学習 | |
[2] 支援技術の概要
私たちは東工大のスーパーコンピュータTSUBAMEの利用支援及び、TSUBAMEを用いることで可能になる大規模分子シミュレーションと構造インフォマティクス技術によるインシリコスクリーニング支援を行っています。
東工大のスーパーコンピュータTSUBAMEは平成18年より運用中のクラスタ型スーパーコンピュータです。計算による科学技術研究の促進を目的として設置され、現在では東工大内のみに限らず学外の研究機関・民間企業の方などに幅広く使われております。平成18年に導入されたTSUBAME1.0 は2度の更新の後、平成22年にTSUBAME2.0に総入れ替えとなり、そして平成25年9月にはTSUBAME2.5へと大幅更新されました。平成29年8月に運用を開始したTSUBAME3.0は、Green500 List(省エネ性能の世界スパコンランキング)において世界1位を達成した上、AI分野で重要な半精度演算性能が47.2ペタフロップスと高く、人工知能分野における需要急増への対応が期待されています。TSUBAME3.0の倍精度の理論演算性能は12.15ペタフロップス(1秒間に12,150兆回の浮動小数点演算が可能)と、スーパーコンピュータ「京」を上回る世界最高レベルの性能となります。最新GPUの採用により、性能および電力効率の向上、ストレージの高速化および大容量化が実現し、計算ノードに搭載されるNVMe対応高速SSDの合算容量は1.08 PBと容量、速度ともに強化され、ビッグデータアプリケーションの処理速度を大幅に加速できます(以上、TSUBAMEを提供する東工大GSICのweb siteから要約)。我々は、このTSUBAME3.0の利用をインシリコユニットを中心に支援しています。
また、TSUBAMEを用いることで可能になる大規模分子シミュレーションを中心とした構造インフォマティクス技術によって、インシリコスクリーニング支援を行っています。具体的には、我々がこれまでに開発した高精度化合物スクリーニング法SIEVE-Scoreやファーマコフォアモデリングの経験、世界最高水準のタンパク質天然変性領域予測システムPrDOS-CNF、大規模並列計算によるタンパク質間相互作用予測MEGADOCKを最大限に活かし、(1)インシリコスクリーニングによるタンパク質と低分子化合物との相互作用様式の推定支援、(2)タンパク質の天然変性領域予測支援、(3)タンパク質間相互作用解析手法に基づく結合候補タンパク質探索と相互作用様式予測支援の3項目の支援を行っています
[3] 支援技術の利用例
TSUBAME3.0上において分子シミュレーションや機械学習に必要な計算機資源、及び各種ソフトウェア(AMBER, Gaussian, GAMESS, LAMMPS, Caffe)を提供しています。
また、TSUBAMEを用いたin silico計算により、熱揺らぎを考慮したドッキングシミュレーションを実施し、バイオアッセイを行うことで阻害活性がある化合物の獲得に成功しています。
[4] 支援担当者の研究概要
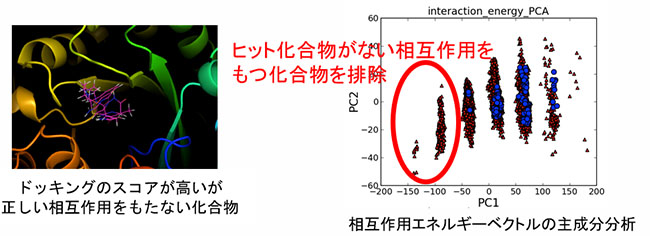
代表者の関嶋は、in silicoのシミュレーションとin vitroの生化学実験を組み合わせた効率的な創薬手法を開発し、20のヒット化合物と、2つの抗原虫活性がありかつ細胞毒性が低い化合物を獲得し、さらに2化合物に対して初めてスペルミジン合成酵素とプトレシン結合部位に化合物が結合した共結晶の立体構造解析(PDBID: 5B1S)に成功しました(生命医薬情報学連合大会 2014 研究奨励賞, R. Yoshino et al., Sci.Rep., 2017)。 インシリコスクリーニング、タンパク質の天然変性領域予測、タンパク質間相互作用解析手法に基づく結合候補タンパク質探索と相互作用様式予測等に関する支援を実施しています。
また、スーパーコンピュータと機械学習を用いた創薬手法の研究を行っており、蛋白質・リガンド間の相互作用をエネルギーベクトルとして機械学習した手法(N. Yasuo and M. Sekijima, JCIM, 2019)においては、Journal of Chemical Information and Modeling誌のMachine Learning Special IssueにおいてSupplementary Cover Artに選ばれた。また、ディープラーニングを用いたヒット化合物予測においては、特許出願も行っている。
天然変性領域は結晶化の妨げとなるため、構造生物学研究者にとって悩みのタネとなっている。分担者の石田が開発した天然変性領域予測システムPrDOSによりタンパク質のアミノ酸配列から予測を行う。本手法は最新の天然変性領域予測に関する国際コンペティション(CASP10 disorder)にて、全28グループ中、最高精度を達成している。
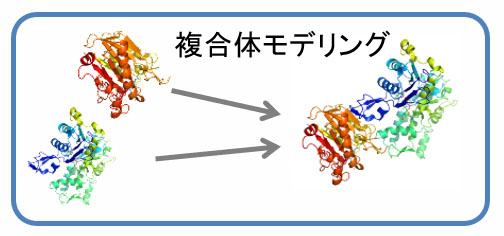
創薬標的としてタンパク質間相互作用が注目を集めているが、相互作用情報の蓄積が不十分なことと複合体構造の決定の難しさが研究上の障壁になっている。結合タンパク質探索と複合体モデリングを実施するソフトウェア分担者の秋山、大上らによって開発されたMEGADOCKにより、標的タンパク質に対して結合するタンパク質の探索と、複合体モデリングを実施している。